近日,动物科技学院张勤教授团队在奶牛行业国际著名期刊《Journal of Dairy Science》在线发表了题为“Longitudinal genome-wide association studies of milk production traits in Holstein cattle using whole-genome sequence data imputed from medium-density chip data”的研究论文。团队博士后滕军和王丹教授为该论文的第一作者,张勤教授和宁超副教授为该论文的通讯作者。
纵向性状(例如奶牛的产奶性状)指的是一个个体有多个表型测量值,并且表型值随时间发生动态变化。每个时间点的测量值称为测定日记录,使用测定日记录分析纵向性状可以最大限度地减少环境噪声,从而提高检验功效。以往奶牛产奶性状的全基因组关联分析研究,要么基于测定日记录(随机回归测定日模型),但是使用的芯片数据;要么使用填充的序列数据,但是基于伪表型(例如女儿产量离差或逆回归育种值;传统动物模型)。
该研究以千牛基因组计划中1059头荷斯坦牛基因型为参考面板,将6470头中国荷斯坦牛的芯片数据填充至全基因组序列数据。然后,基于随机回归测定日模型使用填充的序列数据对产奶性状进行纵向全基因组关联分析。纵向全基因组关联分析共发现了与产奶量、乳脂率和乳蛋白率相关的130个QTL区域。
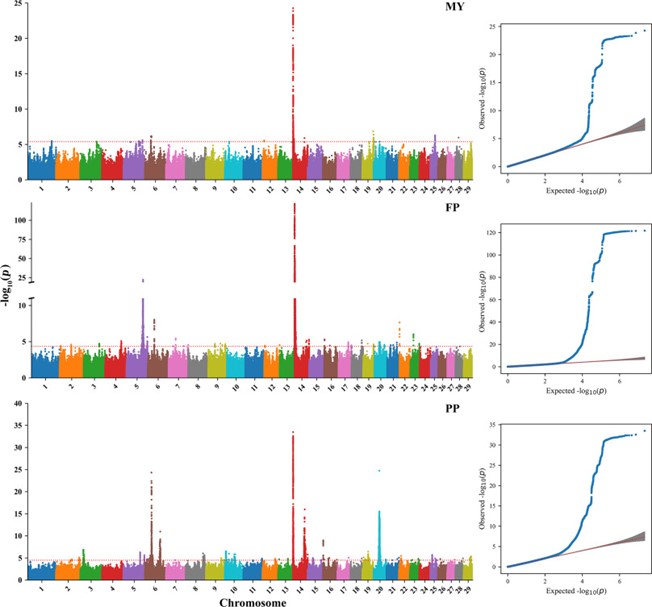
图1. 产奶量、乳脂率和乳蛋白率纵向全基因组关联分析的曼哈顿图和QQ图
为了进一步确定候选基因,该研究使用logP drop的方法估计每个QTL区域的95%置信区间。在这些置信区间中,共发现581个候选基因。此外,该研究重点关注仅包含1个基因或特别显著top SNP的置信区间,在这样的置信区间中发现了28个候选基因。其中大部分已在文献中报道与奶牛产奶性状有关,如DGAT1、HSF1、MGST1、GHR、ABCG2、ADCK5和CSN1S1等。在未报道的基因中,CCSER1、CUX2、SNTB1、RGS7、OSR2和STK3等候选基因显示出良好的潜力,值得进一步研究。
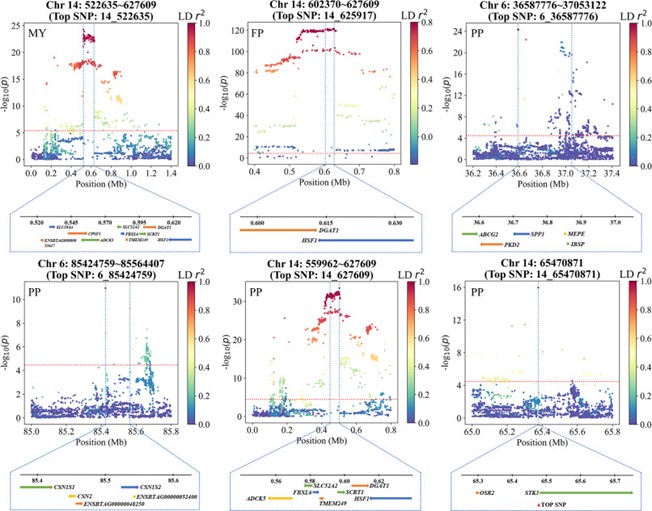
图2. 包含特别显著top SNP的QTL区域的95%置信区间
该研究不仅为产奶性状的候选基因提供了新的见解,而且为使用序列数据基于随机回归测定日模型的纵向全基因组关联分析提供了一般性的分析框架。
该研究得到了国家重点研发计划、扬州大学畜牧学学科特区学科交叉课题、国家自然科学基金、山东省农业良种工程、山东省自然科学基金和国家现代农业产业技术体系的资助。
论文链接:https://doi.org/10.3168/jds.2022-22277
编 辑:万 千
审 核:贾 波









